张琪、许燕团队揭示m6A修饰调控肝脏发育的分子机制
近日,拉斯维加斯lswjs0567官方网站张琪、许燕团队在Nature子刊Nature Communications在线发表了题为“Mettl3-mediated mRNA m6A modification controls postnatal liver development by modulating the transcription factor Hnf4a”的最新研究成果,系统阐述了Mettl3介导的m6A修饰在小鼠出生后肝脏发育成熟中的阶段特异性作用。研究发现,Mettl3介导的m6A修饰是出生后肝脏发育过程必需的,但成年后敲除Mettl3不影响肝脏稳态维持。机制研究发现Mettl3缺失可导致Hnf4a等肝脏核心转录因子转录本上m6A修饰减少,降低Hnf4a mRNA稳定性,最终导致出生后早期小鼠肝脏发育受阻。该研究第一作者为许燕副研究员、科研助理周卓伟(现为密歇根大学博士研究生)和博士研究生康新梅,通讯作者为张琪教授。
肝脏是机体最大的实质器官,也是新陈代谢的中心站。肝脏发育和成熟是一个复杂精细有序的过程,受到胞内外信号、表观遗传调控和转录调控等多层次严密调控[1, 2]。N6-甲基腺苷(m6A)是真核生物mRNA最丰富的修饰,调控转录、核质运输、稳定性和翻译等多个RNA代谢过程。m6A受到甲基化酶和去甲基化酶的动态调控,其中由催化亚基Mettl3和调节亚基Mettl14、 Wtap等组成甲基转移酶复合物负责将m6A加载到转录本上特定位点,去甲基化酶ALKBH5或FTO负责移除m6A修饰[3]。既往研究报道了m6A修饰参与干细胞分化、肿瘤发生等众多生理病理过程[4, 5]。然而,m6A修饰在哺乳动物肝脏发育中的作用尚无系统研究。

图1:文章信息
研究发现,人和小鼠肝脏中m6A修饰水平以及甲基化酶复合物组分Mettl3和Mettl14表达水平在出生后肝脏发育不同阶段动态变化,提示m6A修饰在哺乳动物出生后肝脏发育过程中发挥重要作用。出生前后肝细胞特异性敲除Mettl3(Mettl3flox/flox/Albumin-Cre,Mettl3 cKO)的小鼠出现明显的肝脏脂质沉积、肝细胞凋亡、肝纤维化以及肝前体细胞活化等肝损伤表现,最终在出生后4-7周死亡,说明Mettl3在出生后小鼠肝脏发育中有着不可或缺的作用。
通过m6A-RIP-sequencing和RNA sequencing和一系列验证分析,研究人员发现肝脏中包括Hnf4a在内的一系列肝脏核心转录因子的mRNA上存在m6A修饰。敲除Mettl3后,这些基因转录本上的m6A修饰明显减少,导致Hnf4a的mRNA和蛋白水平及其下游靶标的表达量显著下调。应用腺相关病毒在肝细胞中特异性过表达Hnf4a可以部分缓解Mettl3 cKO小鼠肝损伤,延长存活时间。机制研究发现,敲除Mettl3导致肝细胞中Hnf4a mRNA稳定性下降,这一作用依赖于m6A阅读蛋白Igf2bp1。这些数据说明Mettl3介导的m6A修饰以Igf2bp1依赖的方式促进肝脏核心转录因子Hnf4a mRNA稳定性,维持小鼠出生后肝脏发育。
研究人员进一步探究了Mettl3及其介导的m6A修饰在成体小鼠肝脏稳态维持中的作用。通过构建肝细胞特异性可诱导敲除Mettl3的小鼠(Mettl3flox/flox/Alb-CreERT2,Mettl3 icKO),研究者发现成年后敲除Mettl3不影响小鼠肝脏稳态维持。
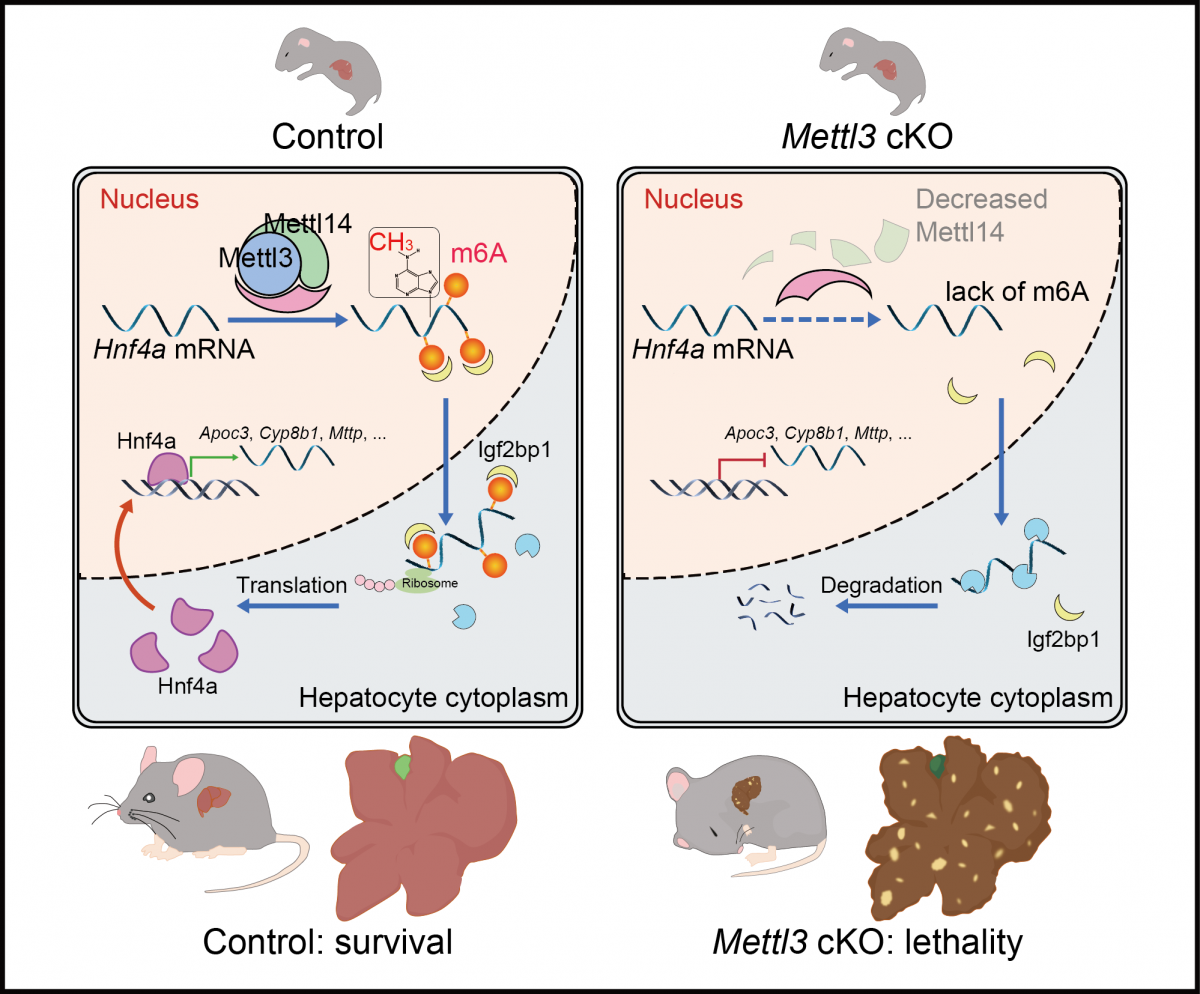
图2:机制图
本研究系统阐述了Mettl3介导的m6A修饰在小鼠出生后肝脏发育成熟中的阶段特异性作用,发现Mettl3介导的m6A修饰是出生后肝脏发育早期必需的,但成年后敲除Mettl3不影响肝脏稳态维持。本研究揭示了肝脏发育成熟的表观遗传调控新机制,为肝脏发育相关重大疾病防治提供了新思路。
张琪、许燕研究团队长期专注于肝脏疾病发生发展的表观遗传调控机制研究及肝脏疾病的细胞治疗研究。在Cell Stem Cell, Journal of Hepatology, Nature Communications, Molecular Therapy等杂志发表多篇文章,先后获得国家重点研发计划、国家自然科学基金面上项目等多个基金支持。
论文链接:https://www.nature.com/articles/s41467-022-32169-4
1. Lau HH, Ng NHJ, Loo LSW, et al. The molecular functions of hepatocyte nuclear factors - In and beyond the liver. J Hepatol 2018;68:1033-1048.
2. Macchi F, Sadler KC. Unraveling the Epigenetic Basis of Liver Development, Regeneration and Disease. Trends Genet 2020;36:587-597.
3. Shi H, Wei J, He C. Where, When, and How: Context-Dependent Functions of RNA Methylation Writers, Readers, and Erasers. Mol Cell 2019;74:640-650.
4. Frye M, Harada BT, Behm M, et al. RNA modifications modulate gene expression during development. Science 2018;361:1346-1349.
5. Liu J, Harada BT, He C. Regulation of Gene Expression by N(6)-methyladenosine in Cancer. Trends Cell Biol 2019;29:487-499.







