张琪、许燕团队揭示Mettl3介导的m6A修饰调控肝纤维化的新机制
近日,拉斯维加斯lswjs0567官方网站生物治疗中心张琪、许燕团队在美国基因与细胞治疗协会(ASGCT)会刊Molecular Therapy上发表了题为“The m6A methyltransferase Mettl3 deficiency attenuates hepatic stellate cell activation and liver fibrosis”的最新研究成果,揭示了Mettl3介导的m6A修饰通过HIPPO/YAP通路调控肝星状细胞活化和肝纤维化进展的新功能,发现靶向Mettl3可以有效抑制肝星状细胞的激活和肝纤维化进程。该研究通讯作者为生物治疗中心张琪教授和许燕副研究员,第一作者为分子医学专业博士研究生李延利和康新梅。
全球遭受肝脏疾病影响的人口近10亿,带来了严重的社会经济负担。我国是肝脏病大国,包括慢性肝炎、脂肪肝和肝硬化在内的慢性肝病患者可能达4.47亿人次,其中超过700万慢性肝病患者发展到了肝硬化阶段(占总人口数0.5%),肝癌患者占全球新发和死亡肝癌患者数目的一半以上[1]。
在各类不同病因引起的慢性肝病中,肝纤维化是共有的进展过程。现代肝脏病学的奠基人、已故肝病学权威Hans Popper教授曾说:“谁能阻止或延缓肝纤维化,谁将能医治大多数慢性肝病”。但目前尚无FDA(美国食品药品监督管理局)或CFDA(国家食品药品监督管理总局)批准的肝纤维化特异性药物。如何在肝纤维化早期阶段及时、及早进行干预,逆转、阻止其进一步恶化,是肝脏疾病研究的热点和难点,也是肝脏疾病治疗的重要突破口。
腺苷酸第六位氮原子上发生的甲基化修饰(N6-methyladenosine, m6A) 是真核生物mRNA最常见的内部修饰。m6A甲基化是由甲基转移酶复合物(Writers)、去甲基化酶(Erasers)和阅读蛋白(Readers)所催化的一个动态可逆修饰。m6A几乎修饰支配着RNA代谢的所有方面(包括mRNA剪接、稳定性、翻译、核质运输和microRNA加工等) ,在各种生理和病理过程中发挥重要作用[2]。在肝脏中,已发现m6A修饰参与脂肪肝、肝脏再生、肝脏缺血-再灌注损伤、肝炎病毒感染、肝内胆管癌和肝细胞癌等各种肝脏疾病的发生发展[3]。然而,m6A修饰在肝纤维化过程中的作用和具体机制尚不清楚。

图1:文章信息
肝星状细胞作为肝脏特有的一种非实质细胞,在肝纤维化进程中发挥着关键作用,其在正常状态下呈静息状态,当肝脏受到物理、化学和微生物感染等病理因素刺激时,肝星状细胞被激活,转变为肌成纤维细胞,并开始大量增殖,成为细胞外基质的主要来源细胞。干预肝星状细胞的活化、增殖和存活有望逆转肝纤维化进程[4]。研究团队通过构建Lrat-Cre介导的肝星状细胞特异性敲除m6A甲基转移酶Mettl3的小鼠(Mettl3 cKO小鼠),发现肝星状细胞特异性敲除Mettl3不影响小鼠发育,但在四氯化碳(CCl4)诱导的肝细胞损伤导致的肝纤维化模型和DDC(3,5-diethoxycarbonyl-1,4-dihydro-collidine)诱导的胆汁淤积导致的纤维化模型中,肝星状细胞特异性敲除Mettl3可以显著降低肝损伤水平、减缓肝纤维化进展。细胞实验也发现敲低/敲除Mettl3显著抑制肝星状细胞的活化和纤维化基因表达。这些结果表明靶向Mettl3可有效抑制肝星状细胞活化和肝纤维化进展,进一步的机制探究证实Hippo/YAP信号通路在其中发挥着重要作用,为肝纤维化早期干预和药物研发提供了新靶标。
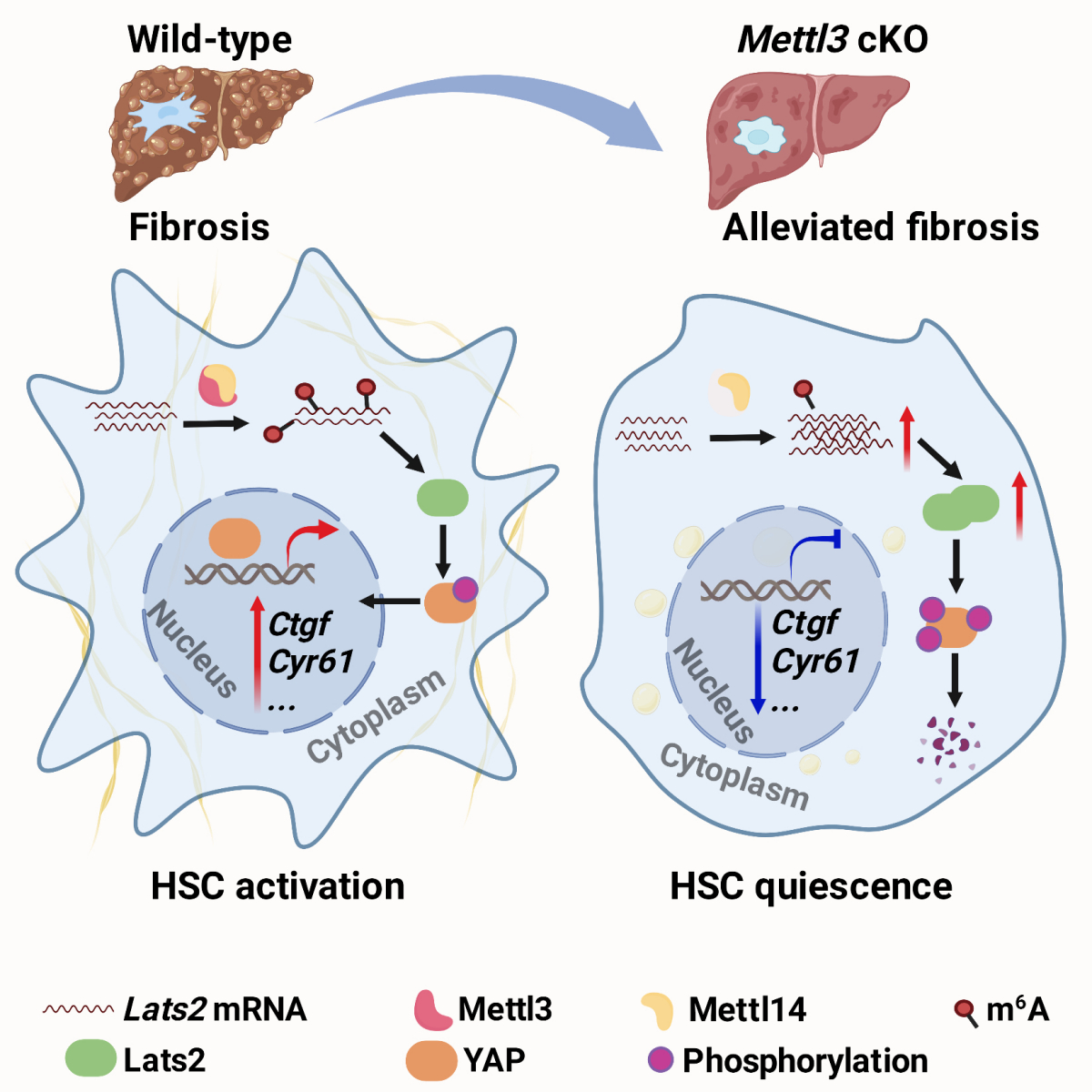
图2:Mettl3介导的m6A修饰调控肝星状细胞活化及肝纤维化的机制示意图
张琪、许燕研究团队长期专注于肝脏疾病发生发展的表观遗传调控机制研究及肝脏疾病的细胞治疗研究,在Cell Stem Cell, Journal of Hepatology, Nature Communications, Molecular Therapy等杂志发表多篇文章,并获得国家重点研发计划、国家自然科学基金面上项目等基金支持。
论文链接:https://www.cell.com/molecular-therapy-family/molecular-therapy/fulltext/S1525-0016(22)00441-5
参考文献:
1. Xiao J, Wang F, Wong NK, et al. Global liver disease burdens and research trends: Analysis from a Chinese perspective. J Hepatol 2019;71:212-221.
2. Zhao BS, Roundtree IA, He C. Post-transcriptional gene regulation by mRNA modifications. Nat Rev Mol Cell Biol 2017;18:31-42.
3. Zhao Z, Meng J, Su R, et al. Epitranscriptomics in liver disease: Basic concepts and therapeutic potential. J Hepatol 2020;73:664-679.
4. Tsuchida T, Friedman SL. Mechanisms of hepatic stellate cell activation. Nat Rev Gastroenterol Hepatol 2017;14:397-411.







